Autor: Peter Pančík
Publikované dňa:
Citácia: PANČÍK, Peter. 2020. Biopedia.sk: O pôvode nového koronavírusu (SARS-CoV-2). [cit. 2024-04-25]. Dostupné na internete: <https://biopedia.sk/virusy-a-bakterie/o-povode-noveho-koronavirusu-sars-cov-2>.
SARS–CoV–2 (predtým 2019–nCoV) je nový koronavírus, pôvodne identifikovaný v ľudskej populácii čínskeho mesta Wuhan (provincia Hubei), u ktorej sa prejavili symptómy závažného respiračného ochorenia, medzičasom pomenovaného ako COVID–19 (COronaVIrus Disease–2019).(1)(2)(3) Odtiaľ sa ochorenie rozšírilo do celého sveta. Svetová zdravotníctva organizácia (WHO) 30.1.2020 vyhlásila v súvislosti s nákazou ohrozenie verejného zdravia medzinárodného významu. Ku dňu publikovania tohto článku (3.5.2020) bolo podľa WHO celosvetovo identifikovaných 3 267 184 prípadov nákazy, z ktorých 229 971 skončilo smrťou (7%).
Taxonómia, morfológia a genóm link
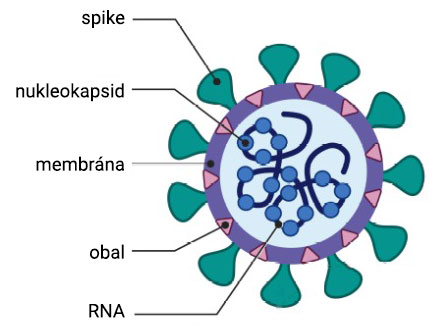
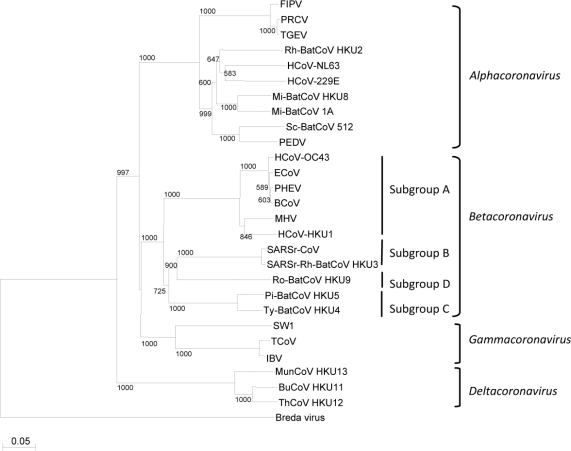
Koronavírusy (čeľaď Coronaviridae, rad Nidovirales) sú patogény ľudí a zvierat. Sú to obalené vírusy, ktorých genóm je tvorený jednovláknovou RNA pozitívnej polarity (+ssRNA) o veľkosti 26–32 kb.(4) Rozdeľujú sa do 4 rodov: α– a β–koronavírusy infikujú cicavce, δ– a γ–koronavírusy predovšetkým vtáky a niektoré cicavce.(5) Nový koronavírus, tak ako aj vírusy, ktoré spôsobili závažné epidémie SARS (Severe Acute Respiratory Syndrome) a MERS (Middle–East Respiratory Syndrome) v roku 2003, resp. 2012, zaraďujeme medzi zoonotické (živočíšneho pôvodu) β–koronavírusy.(6)(7)(8)
Genóm SARS–CoV–2 pozostáva z 29903 nukleotidov. Na oboch koncoch obsahuje neprekladané oblasti, tzv. UTR (untranslated region), a 11 otvorených čítacích rámcov (ORF), ktoré kódujú 27 proteínov. Podobnosť genómu SARS–CoV–2 (NCBI Reference Sequence: NC_045512.2) s genómom SARS–CoV (NC_004718.3) je 88%, s genómom MERS–CoV iba 50%. Najväčšia podobnosť je práve s rôznymi SARS–u podobnými koronavírusmi netopierov (až 96%).(9)
Prekonanie medzidruhovej bariéry link
Rozhodujúcim predpokladom infekcie hostiteľa je špecifická interakcia niektorého z povrchových proteínov vírusu a receptora hostiteľskej bunky. Absencia tejto špecifickej interakcie predstavuje bariéru pre schopnosť vírusu nakaziť daný organizmus. Mutácie sú však zdrojom prirodzenej genetickej variability, a tak s určitou pravdepodobnosťou môže dôjsť k vytvoreniu takej varianty vírusového proteínu, ktorý rozozná príslušný receptor u hostiteľa.
Miera schopnosti mutovať a prekonávať medzidruhové bariéry ako aj prispôsobivosť na odlišné typy tkanív je u koronavírusov obzvlášť vysoká.(10)(11) Rekombinácia je ďalším z mechanizmov evolúcie u koronavírusov, vrátane SARS–CoV a MERS–CoV.(12)(13)(14) Ohľadom rekombinácie u SARS–CoV–2 sú dôkazy zatiaľ nejednoznačné.(15)(16)
Práve netopiere sa ukazujú ako prírodný rezervoár koronavírusov,(17)(18) ale až dokiaľ v roku 2003 nevypukla epidémia SARS, sa o týchto vírusoch a ich prenose na človeka vedelo len veľmi málo. Výskum koronavírusov, ako aj biológie samotných netopierov, od tej doby veľmi pokročil.
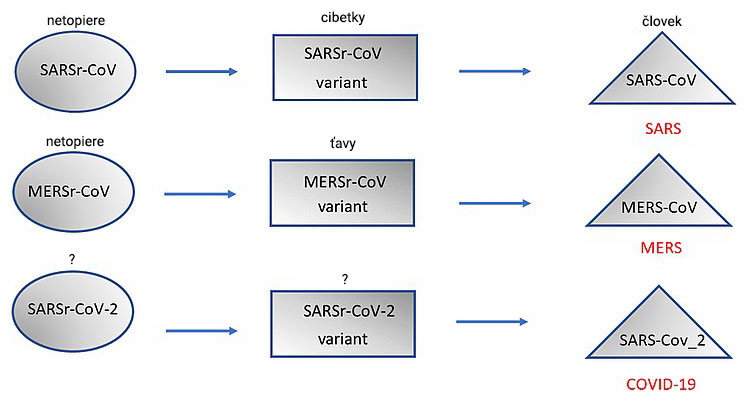
V prípade epidémie SARS a MERS však zrejme nedošlo k priamemu prenosu vírusu z netopierov na ľudí, ale prostredníctvom tzv. medzihostiteľa, akým boli cibetky (SARS(19)) a ťavy (MERS(20)). Hoci je pôvod vírusu SARS–CoV–2 ako aj jeho medzihostiteľ doposiaľ neznámy, predchádzajúce epidémie príbuzných koronavírusov ako aj porovnávacie štúdie naznačujú, akou cestou sa vo výskume uberať.
„Spike“ proteín link
Zistilo sa, že rozhodujúcim faktorom pri schopnosti SARS–CoV a MERS–CoV infikovať človeka, je špecifická väzba tzv. spike glykoproteínu (S) vírusu s receptorom ACE2 (angiotensin-converting enzyme 2),(22) resp. dipeptidyl peptidázou 4 (DPP4; tiež CD26).(23) Oba receptory sa nachádzajú v špecifických bunkách epitelovej výstelky priedušiek a pľúcnych alveolách u ľudí.
SARS–CoV–2 taktiež využíva pre vstup do bunky ACE2 receptor,(9) avšak podobnosť kľúčovej rozpoznávajúcej aminokyselinovej sekvencie je pri týchto dvoch vírusoch veľmi nízka.(24) Z bioinformatickej analýzy dokonca vyplýva, že spike proteín s takto zmenenou štruktúrou by nemal mať schopnosť účinne interagovať s ACE2 receptorom. Realita je však odlišná, čoho príčinou je pravdepodobne vznik novej „optimálnej“ interakcie medzi spike proteínom SARS–CoV–2 a ACE2 receptorom. To by zároveň malo vyvrátiť hypotézu, že by nový koronavírus bol účelne nadizajnovaný v laboratóriu, pretože nový spôsob interakcie, a v konečnom dôsledku vysoká afinita k ACE2 receptoru, nemohla byť predpovedaná dopredu.(24)
Novinkou sú však aj O–viazané oligosacharidy a polybázické štiepne miesto, ktoré u SARS–u podobných koronavírusov úplne chýbajú, vyskytujú sa však u niektorých iných koronavírusov, napr. aj HCoV–OC43, MERS–CoV, MHV–A59.(25) Prítomnosť tejto novej sekvencie vytvára podmienky pre zvýšenú virulenciu(26) a zväčšenie rozsahu bunkových typov, aké vírus dokáže infikovať.(27) Nový motív vytvára štiepne miesto medzi S1/S2 podjednotkou spike proteínu prostredníctvom furín proteázy, čo uľahčuje vstup vírusu do bunky, predovšetkým v pľúcach.(28)
Pátranie po pôvode vírusu link
Istá čínska skupina virológov napísala manuskript o tom, že SARS–CoV–2 obsahuje sekvencie z HIV, čo by podporilo hypotézu o umelom vytvorení vírusu. Bioinformatickou analýzou sa neskôr ukázalo, že sekvencie nie sú HIV–1 špecifické, a že sa nachádzajú aj u ostatných koronavírusov.(29) Manuskript bol napokon stiahnutý.
V súvislosti s výskumom SARS–u podobného vírusu netopierov (RaTG13–CoV) priamo vo Wuhane sa taktiež uvádza, že by nový koronavírus mohol pochádzať práve z tohto laboratória, príp. byť priamo manipuláciami odvodeným od tohto vírusu. Genómová sekvencia SARS–CoV–2 a RaTG13–CoV sa zhoduje v približne 96,3%, pričom mutácie sú roztrúsené po celom genóme15. Rozdiel je však aj práve v spomínanej kľúčovej sekvencii spike proteínu.(25) Naproti tomu napr. zhoda ľudského SARS-CoV s vírusom medzihostiteľa – cibetiek – je až 99,8%.(30)
Prvotné výskumy poukazovali okrem spojitosti SARS–CoV–2 s netopiermi ešte aj na podobnosť s hadmi,(31) avšak použitá metodika je pri identifikovaní pôvodného hostiteľa veľmi sporná.
V neposlednom rade sa za možného pôvodcu SARS–CoV–2 považuje zástupca šupinavcov (pangolin), ktorý je súčasťou tradičnej čínskej medicíny, hoci je zákonom chránený a v Číne hrozí za jeho predaj väzenie. Túto hypotézu podporuje aj fakt, že aktívna doména spike glykoproteínu SARS–CoV–2 vykazuje vysokú homológiu práve s vírusom identifikovaným u tohto živočícha, pričom celogenómová zhoda týchto dvoch vírusov je až 99%.(24)(32)
Časť vedcov sa nestotožňuje s myšlienkou, že pôvodným miestom, odkiaľ došlo k šíreniu vírusu v ľudskej populácii, je trhovisko vo Wuhane, pretože nie všetky prípady infikovaných ľudí mali prepojenie na trhovisko. Taktiež následné kroky, jedným z nich aj zatvorenie trhoviska 1.1.2020, znemožnili identifikovať pôvodného šíriteľa, ak taký na mieste existoval.(33)(16)(3)
Dopátrať sa k pôvodnému hostiteľovi je v prípade SARS–CoV–2 o to ťažšie, že v mnohých prípadoch je vírus prenášaný prostredníctvom superšíriteľov, ktorí sami o sebe nevykazujú žiadne známky ochorenia. Taktiež ochorenie u časti populácie prebieha len s miernymi príznakmi, takže mnohé z prípadov ostanú neidentifikované.(16)
Pár slov na záver... link
Súčasťou virologického výskumu je aj cielená mutagenéza, pretože nám umožňuje študovať funkciu jednotlivých génov a ich proteínov a tým porozumieť mechanizmom infekcie a účinne navrhovať terapeutické ciele. Nové techniky génového inžinierstva umožňujú vytvárať nové varianty a kombinácie génov za laboratórnych podmienok.(18)
Na záver možno predsa len trošku vlastnej úvahy. Viem si predstaviť, že postupnou „skladačkou“ a cielenými zámenami nukleotidov v kľúčových oblastiach možno vytvoriť variantu vírusu s požadovanými vlastnosťami. Do hry však, okrem našich doterajších znalostí, vstupuje viac premenných, vrátane času, koľko by daný vírus musel byť pasážovaný na bunkových kultúrach, čím by sa im prispôsobil natoľko, že by v podstate znížil svoju schopnosť prežívania v prírode (tzv. fitness). Nový koronavírus však naopak disponuje vlastnosťami, ktoré nie sú úplne typické pre „laboratórne“ vírusy.
Dôležité je zdôrazniť, že aj keď je teória o úniku z laboratória považovaná za menej pravdepodobnú, vedci súhlasia, že by sa mala ďalej skúmať ako súčasť úsilia o pochopenie pôvodu SARS–CoV–2. Bez definitívnych dôkazov zostáva otázka pôvodu vírusu otvorená.
- Chan, Jasper Fuk-Woo, et al.: A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. (2020). The Lancet 395.10223: 514-523.
- Yang, Xiaobo, et al.: Clinical course and outcomes of critically ill patients with SARS-CoV-2 pneumonia in Wuhan, China: a single-centered, retrospective, observational study. (2020). The Lancet Respiratory Medicine.
- Huang, Chaolin, et al.: Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. (2020). The Lancet 395.10223: 497-506.
- Weiss, Susan R., and Sonia Navas-Martin: Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus. (2005). Microbiol. Mol. Biol. Rev. 69.4: 635-664.
- Woo, Patrick CY, et al.: Discovery of seven novel Mammalian and avian coronaviruses in the genus δ-coronavirus supports bat coronaviruses as the gene source of α-coronavirus and β-coronavirus and avian coronaviruses as the gene source of γ-coronavirus and δ-coronavirus. (2012). Journal of virology 86.7: 3995-4008.
- Drosten, Christian, et al.: Identification of a novel coronavirus in patients with severe acute respiratory syndrome. (2003). New England journal of medicine 348.20: 1967-1976.
- Zaki, Ali M., et al.: Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. (2012). New England Journal of Medicine 367.19: 1814-1820.
- Wan, Yushun, et al.: Receptor recognition by the novel coronavirus from Wuhan: an analysis based on decade-long structural studies of SARS coronavirus. (2020). Journal of virology 94.7.
- Zhou, Peng, et al.: A pneumonia outbreak associated with a new coronavirus of probable bat origin. (2020). Nature 579.7798: 270-273.
- Decaro, Nicola, et al.: Recombinant canine coronaviruses in dogs, Europe. (2010). Emerging infectious diseases 16.1: 41.
- Elena, Santiago F., and Rafael Sanjuán: Adaptive value of high mutation rates of RNA viruses: separating causes from consequences. (2005). Journal of virology 79.18: 11555-11558.
- Lau, Susanna KP, et al.: Molecular epidemiology of human coronavirus OC43 reveals evolution of different genotypes over time and recent emergence of a novel genotype due to natural recombination. (2011). Journal of virology 85.21: 11325-11337.
- Zhang, X. W., Y. L. Yap, and A. Danchin: Testing the hypothesis of a recombinant origin of the SARS-associated coronavirus. (2005). Archives of virology 150.1: 1-20.
- Zhang, Zhao, Libing Shen, and Xun Gu: Evolutionary dynamics of MERS-CoV: potential recombination, positive selection and transmission. (2016). Scientific Reports 6.1: 1-10.
- Helmy, Yosra A., et al.: The COVID-19 Pandemic: A Comprehensive Review of Taxonomy, Genetics, Epidemiology, Diagnosis, Treatment, and Control. (2020). Journal of Clinical Medicine 9.4: 1225.
- Yu, Wen-Bin, et al.: Decoding the evolution and transmissions of the novel pneumonia coronavirus (SARS-CoV-2) using whole genomic data. (2020). ChinaXiv 202002.v2: 5.
- Li, Wendong, et al.: Bats are natural reservoirs of SARS-like coronaviruses. (2005). Science 310.5748: 676-679.
- Cui, Jie, Fang Li, and Zheng-Li Shi: Origin and evolution of pathogenic coronaviruses. (2019). Nature reviews Microbiology 17.3: 181-192.
- Guan, Yi, et al.: Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China. (2003). Science 302.5643: 276-278.
- Alagaili, Abdulaziz N., et al.: Middle East respiratory syndrome coronavirus infection in dromedary camels in Saudi Arabia. (2014). MBio 5.2: e00884-14.
- Corman, Victor M., et al.: Hosts and sources of endemic human coronaviruses. (2018). Advances in virus research. Vol. 100. Academic Press. 163-188.
- Li, Wenhui, et al.: Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. (2003). Nature 426.6965: 450-454.
- Lu, Guangwen, et al.: Molecular basis of binding between novel human coronavirus MERS-CoV and its receptor CD26. (2013). Nature 500.7461: 227-231.
- Andersen, Kristian G., et al.: The proximal origin of SARS-CoV-2. (2020). Nature medicine 26.4: 450-452.
- Coutard, Bruno, et al.: The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. (2020). Antiviral research 176: 104742.
- Claas, Eric CJ, et al.: Human influenza A H5N1 virus related to a highly pathogenic avian influenza virus. (1998). The Lancet 351.9101: 472-477.
- Kido, Hiroshi, et al.: Role of host cellular proteases in the pathogenesis of influenza and influenza-induced multiple organ failure. (2012). Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics 1824.1: 186-194.
- Hoffmann, M., Kleine-Weber, H. & Pöhlmann, S: A multibasic cleavage site in the spike protein of SARS-CoV-2 is essential for infection of human lung cells. (2020). Mol Cell, in press, doi:DOI: 10.1016/j.molcel.2020.04.022.
- Xiao, Chuan, et al.: HIV-1 did not contribute to the 2019-nCoV genome. (2020). Emerging Microbes & Infections 9.1: 378-381.
- Song, Huai-Dong, et al.: Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human. (2005). Proceedings of the National Academy of Sciences 102.7: 2430-2435.
- Ji, Wei, et al.: Cross‐species transmission of the newly identified coronavirus 2019‐nCoV. (2020). Journal of medical virology 92.4: 433-440.
- Cyranoski, David: Did pangolins spread the China coronavirus to people?. (2020). Nature.
- Cohen, Jon: Wuhan seafood market may not be source of novel virus spreading globally. (2020). Science 10.